Después de haber estudiado los comandos básicos de python, las estructuras y lo esencial para poder realizar cualquier código de programación, como consejo para todos los lectores, es que antes de programar es entender bien el problema que se va a resolver y después lo pasamos al lenguaje python.
En esta lección aprenderemos a usar los textos enfocados en problemas de biología.
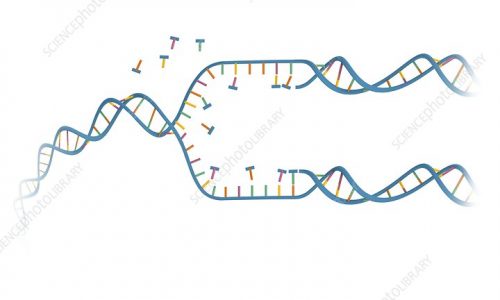
La reproducción del genoma es una de las tareas más importantes antes de dividirse. Antes de que una Célula de divida esta se reproduce, para que cada célula hija obtenga una copia de la información genética. Como resultado se empieza con un par de cadenas complementarias de ADN y se termina con 2 pares complementarias.
Vamos a empezar por encontrar el ori de algunos genomas de bacterias, las cuales tienen cientos de nucleótidos, el motivo de esto es econtrar el ori en algunas otras bacterias (Cabe resaltar que los ejemplos usados, son solo ejemplos y algunos pueden ser inventados, solo con el fin de practicar). El siguiente ejemplo es de la bacteria vibrio-cholerae, el tiene la siguiente secuencia de nucleótidos:

Dentro de esta secuencia de nucleótidos, hay un patrón qque es el que más se repite, lo llamremos K-mer. Con ayuda de la informática y lo aprendido en python analizaremos la base de nucletidos para obtener cual es el patrón que más se repite
Pot ejemplo, “ACTAT” es una cadene frecuente en:
ACAACTATGCATACTATCGGGAACTATCCT
Para poder realizar está busqueda es necesario tener un contador, que en cada vuelta hará un recorrido en las bases nitrogenadas dependiendo de cuantos caracteres tenga en patrón:
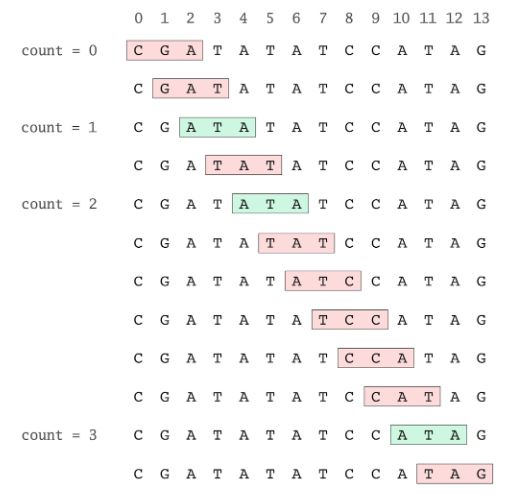
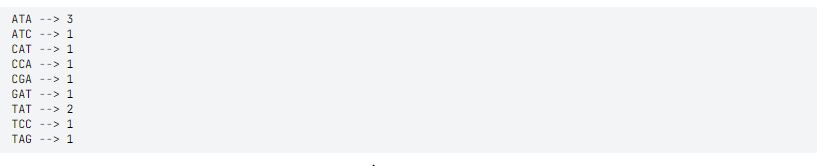
Por ejemplo: Queremos saber cuales son los k mer más frecuentes en un texto -Text = “CGATATATCCATAG”- and k=3, crearemos una función que nos ayude hacer este conteo.
Recuerde que los nucleótidos A y T son complementos entre sí, al igual que C y G. Teniendo una hebra de ADN y un suministro de nucleótidos “flotantes libres” como se muestra en la figura a continuación, podemos imaginar la síntesis de una hebra complementaria en un hilo de plantilla. La siguiente figura muestra una hebra molde “AGTCGCATAGT” y su hebra complementaria “ACTATGCGACT”.
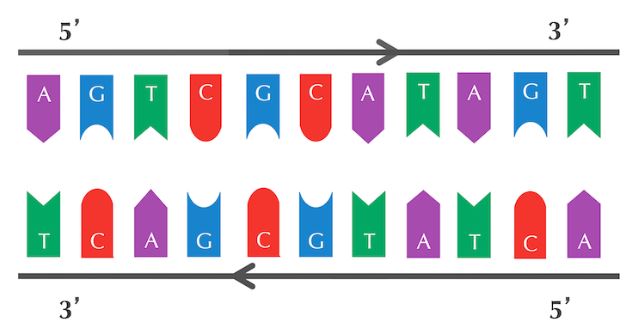
L a hebra complementaria en la figura a continuación se lee “TCAGCGTATCA” de izquierda a derecha en lugar de “ACTATGCGACT”. A y T son complementarios entre sí, al igual que C y G. El comienzo y el final de una hebra de ADN se denotan 5′ (se pronuncia “cinco primos”) y 3′ (se pronuncia “tres primos”), respectivamente. Cada hebra de ADN se lee en dirección 5′ → 3′, y la hebra complementaria corre en dirección opuesta a la hebra molde. Consulte DESVÍO: Direccionalidad de las cadenas de ADN para saber por qué los biólogos usan 5′ y 3′ para referirse al principio y al final de una cadena de ADN.